抗体由来N-結合型糖鎖の構造同定
抗体由来N-結合型糖鎖の構造同定
高精度多段階MSnスペクトルにより糖鎖構造異性体を判別
抗体から切り出して得たPA化糖鎖の糖鎖微量迅速解析システム2による構造同定例を示します。PA化糖鎖は,Human Myeloma IgG1抗体(5ug)のSDS-PAGEゲル切片から酵素(PNGase F)によってN-結合型糖鎖を切り出し,シアリダーゼ処理した後,PAラベル化・精製し調製しました。
PA化糖鎖は液体クロマトグラフProminence nano®で分離し,MALDIプレート用スポッティング装置Accuspot®でMALDIプレートにマトリックス溶液とともにスポットしました。
マトリックスにはDHB(2,5-dihydroxybenzoic acid)を用いています。
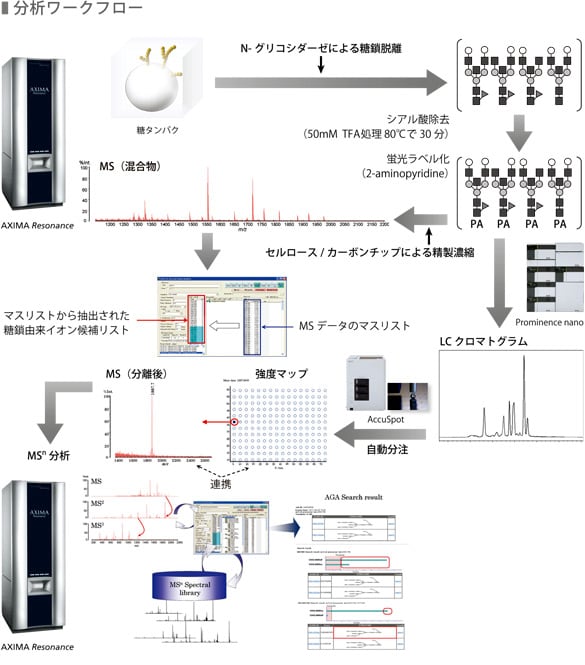
糖鎖微量迅速解析システム2(Accurate Glycan Analyzer 2)では,MALDI 質量分析計を用いることの利点を生かし,糖鎖混合物状態でのマススペクトルから糖鎖由来のイオンをピックアップすることができます。
このことにより,LC 分離後のイオン探索が容易に行えます。
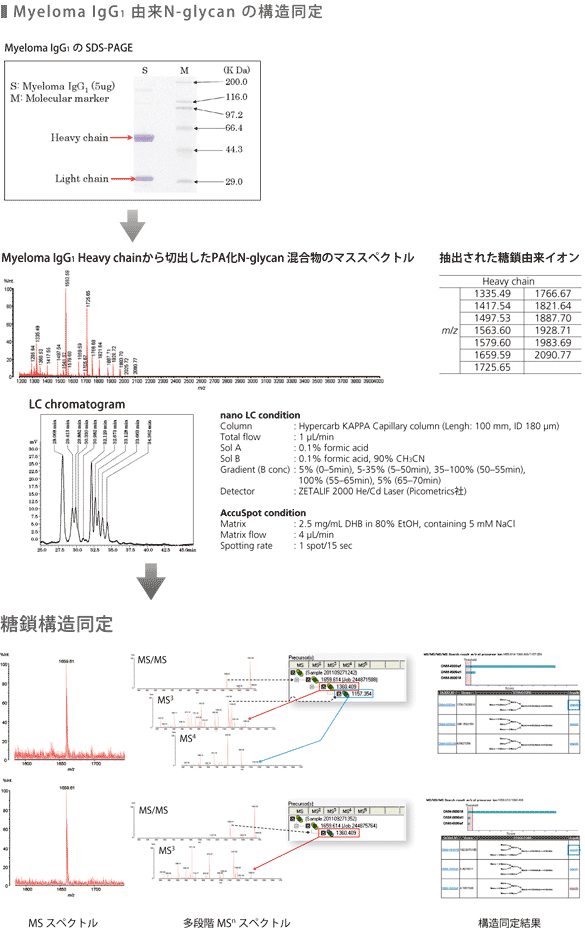
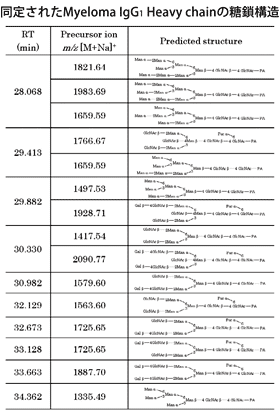
Myeloma IgG1Heavy Chainにて同じm/z 1659.6を示すLC溶出時間28.1minと29.4minに含まれるPA化糖鎖の構造同定例を示します。両者ともに同じm/zを持っており,両者のMS/MSスペクトルを比較してもスペクトルパターンに違いは見られませんが,データベースからクライアント2を通して提示されたm/z1360.4のプロダクトイオンをプリカーサイオンとして測定したMS3スペクトルではm/z 874.3,m/z 671.2などいくつかのプロダクトイオンの相対的な信号強度に差が見られました。このMS3スペクトルデータを使ってデータベース検索することにより,構造異性体を識別することができました。構造によってはさらにMS4プリカーサーイオンが提示され,MS4スペクトルを使用したデータベース検索により構造を同定します。
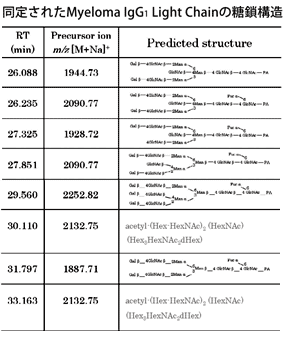
このようにして同定されたMyeloma IgG1のHeavy chainとLight chainそれぞれの糖鎖構造を表に示しました。