MicrobialTrack™ - 特長
MALDI-TOFMS 微生物同定ソフトウェア
ゲノム情報から構築されたデータベース
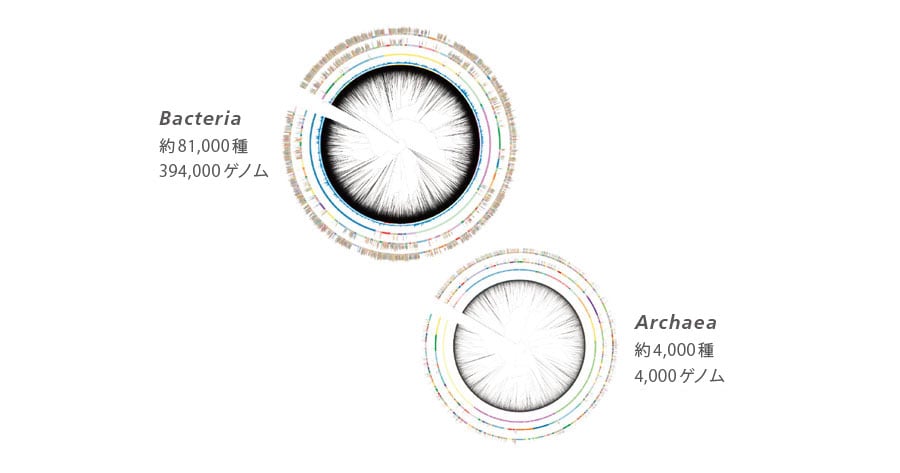
原核微生物(バクテリア、アーキア)の既知種 約1万8千種に加えて、ゲノムからその存在が示唆される難培養・未培養微生物を含む約8万5千種を登録したデータベースを開発しました。このデータベースにより、従来のMALDI-TOFMS 微生物同定システムを超えた広範な系統分類群の同定が可能になります。
遺伝子解析では、培養完了から微生物種が同定されるまで96検体で2日程度必要でしたが、MicrobialTrackとMALDI-TOFMSを利用すれば、3時間以下注)で同定を終えることができます。
注)液体培養した資料微生物株を、有機酸と有機溶媒とを用いて前処理を行った場合
ゲノム配列の類似度に基づく種の定義
同定結果として表示される微生物の学名は、Genome Taxonomy Database(GTDB release 214, gtdb.ecogenomic.org/stats/r214)に準拠しています。GTDBにおける種の定義は、菌株間のゲノム配列の類似度をコンピュータ上で計算することで微生物同定を行う方法に依拠しており、そのaverage nucleotide identity(ANI:2つのゲノム配列間で共通する相同領域の相同性%)およびalignment factor(AF:2つのゲノム配列間で共通する相同領域の割合%)に基づきます。すなわち、GTDBにおける分類体系では、同種の基準は原則95%以上のANI、65%以上のAFで規定されています。本分類体系では、属レベルの分類にも同様のゲノム配列の比較に依拠した基準が適用されています。
MicrobialTrackのデータベースには、未培養および難培養微生物などに由来するゲノム情報、すなわち環境メタゲノム情報やシングルセルゲノム情報などに由来するタンパク質の理論質量情報も含まれます。これら未培養微生物群には正式な学名が与えられていないことが多く、そのような場合、GTDBでは属種名等が記号で記載されていることがあります(例えばMGIIa-L1やsp002965065など)。これらについては、当該分類群を示す記号であり、正式な学名ではないことにご注意ください。これらの記号で記述されている分類群は、GTDBホームページでその詳細を確認することが可能です(https://gtdb.ecogenomic.org)。これら分類群は、今後の研究の進展により正式な学名が与えられ、名称が変更される可能性があります。
目的に応じて使い分けられる2種類のデータベース
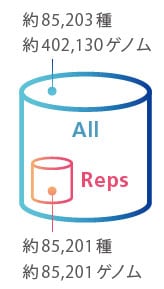
- すべてのゲノム配列に由来する質量情報を登録したデータベース(All)と、種レベル分類群の代表ゲノム配列情報のみから推定した理論質量を登録したデータベース(Representatives:Reps)をご利用いただけます。
- 本データベースには、種レベルの分類群中に複数株に由来するゲノム配列情報から推定された理論質量情報が含まれる場合があります(例えば、Escherichia coliで3万を超えるゲノム配列情報からの推定されたタンパク質質量情報が格納されています)。Allはそのすべての情報を対象にした検索を実施します。一方、Repsは種レベル分類群を代表する1件のゲノム配列情報からの質量情報を格納したデータベースを対象に検索を行います
- Allを使用すれば、株レベルで類似度の高い菌株(由来ゲノム配列情報)の選抜に役立ちます。
- Repsは迅速に種レベルでの分類群を推定する際に便利です。
- 類似度が高い他の種レベル分類群を確認したい場合にも、Repsを用いる検索が便利です。
注:データ数は2025年3月時点の情報です。
定期的アップデート

本データベースは、定期アップデートにより、ゲノム配列情報の蓄積や新しい分類群の追加など、分類体系のダイナミズムにもフレキシブルに対応します。利用期間内であれば、定期アップデート後のデータベースは追加の費用や手続きなしでご利用いただけます。
プロテオミクスによる微生物同定技術
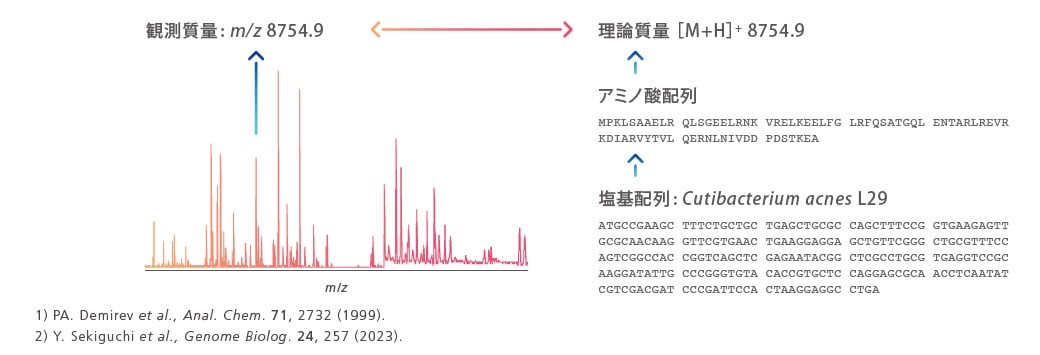
MicrobialTrackは、ゲノム情報に基づいて推定されたタンパク質の理論質量データベースを活用します。微生物由来のタンパク質の観測質量と理論質量が一致することで、その微生物種を同定できると考えられています1)。近年のゲノム解析技術の進展により、公共の塩基配列データベースには膨大な原核微生物のゲノム情報が蓄積されています。この情報を基に開発された大規模なタンパク質の理論質量データベースと独自に開発した同定アルゴリズムにより、培養条件や試料調製法の影響を受けることなく、多様な微生物種を迅速に同定することが可能になりました2)。
どこからでもアクセス可能なクラウドサービス
場所を選ばず、導入も簡単
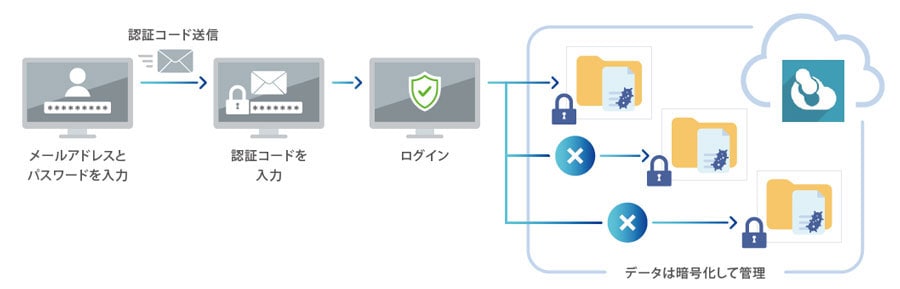
クラウドサービスのため、専用PCの購入やソフトウェアのインストールは不要です。インターネットに接続できる環境さえあれば、実験室や居室など場所を問わず、お手持ちのPCから指定のURLにアクセスしてご利用いただけます。
万全のセキュリティ体制
本サービスは、信頼性の高いクラウド基盤を活用し、アクセスコントロールを含む厳格なセキュリティ対策のもとで運用されています。定期的なアクセスログの監視や脆弱性診断、ソフトウェア更新を通じて常に安全性を維持し、他の利用者のデータが参照されない設計を採用しているため、安心してご利用いただけます。さらに、多要素認証(MFA)をご活用いただくことで、より一層の安全性を確保できます。
簡便なソフトウェア操作性
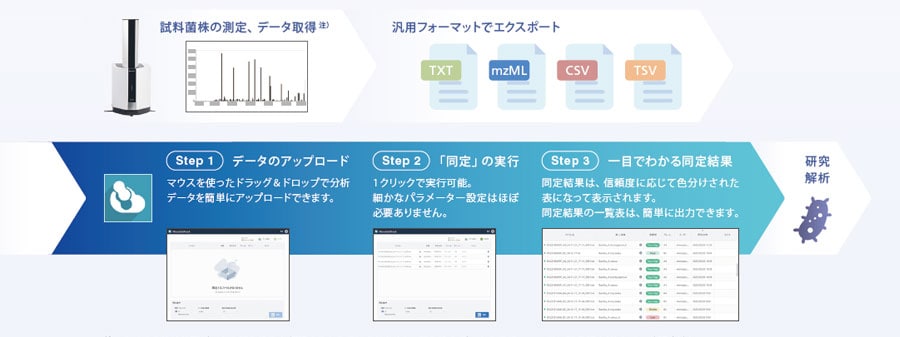
わずか3ステップで同定完了
微生物をMALDI-TOFMSで測定します。汎用フォーマットに対応しているため、データを取得する装置に制限はありません。得られたピークリストを理論質量データベースに照合して微生物を同定します。同定は、わずか3ステップで行うことができます。
同定結果は、信頼度に応じた色付きアイコンとともに候補微生物種がランキング形式で表示され、ピークが微生物種のどのタンパク質にヒットしたかの帰属情報も確認できます。
注:すべての他社装置データの読み込みを保証するものではありません。読み込みデータの実績については製品紹介Webでご確認ください。
識別困難な微生物群 “コンプレックス”
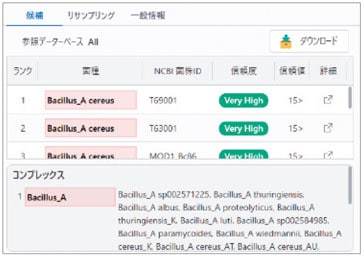
MicrobialTrackでは、菌株間のゲノム配列の類似度を基に種を定義します。具体的には、average nucleotide identity(ANI:2つのゲノム配列間の相同領域の相同性%)95%以上と、alignment factor(AF:共通する相同領域の割合%)65%以上を基準としています。
しかし、定義上は別種でもタンパク質の理論質量の一致度が高く、マススペクトルのみでは識別が難しい微生物分類群が存在します。
MicrobialTrackでは、技術的に識別困難なそれらの微生物群を“コンプレックス”と定義しています。理論質量の類似度から“コンプレックス”を特定し、分かりやすくお知らせします。
同定結果の多面的評価

同定結果は、試料微生物の状態や試料調製、およびそれらが反映されたMALDIマススペクトルの品質などの要因によって変動します。
そのため、本製品では各同定結果を多面的に評価するために複数の指標を導入しています。
そのうちの1つとして、観測ピークの一部をランダムに選択して繰り返し同定を行い、第一候補として推定される属種がどの程度の頻度で推定されたかを示す手法を採用しています。これにより、属レベル・種レベルでの推定結果を客観的に確認できます。
ピークが帰属されたタンパク質情報

観測された各ピークが、どのタンパク質に由来するかを理論質量情報から推定し、グラフと表で表示します。ゲノム情報から推定された理論タンパク質質量と一致した質量ピークは赤く表示されるため、一目で確認できます。
オプション機能では、それらのタンパク質の名称や理論質量、アミノ酸配列なども確認できます。


