細胞培地分析プラットフォーム C2MAPシステム - 特長
細胞培地分析プラットフォーム
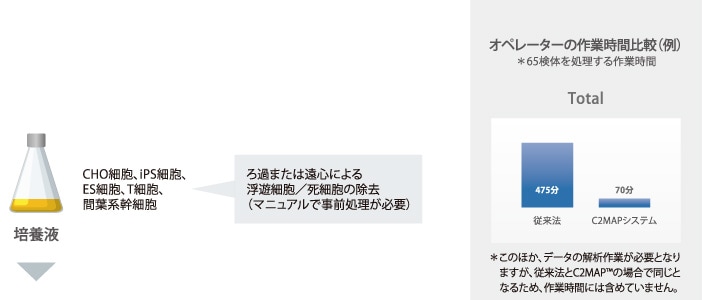
培養上清分析の前処理から測定までを自動化
- 前処理から測定までの自動化により、夜間や休日の自動分析が可能
- 実際の培養にあわせた測定ワークフローが選択可能
- 前処理装置からLC/MS/MS装置まで、シームレスな分析と管理が可能
- 前処理後の試料はマイクロプレートにストックされるため、再測定が容易


幅広い測定化合物・培養上清試料に対応
- 動物細胞用の主要基礎培地成分および分泌代謝物、計95成分の高速一斉分析が可能
- 広範な細胞培地での実績(iPS細胞、ES細胞、間葉系幹細胞、T細胞、CHO細胞)
制御SWに収録されている分析メソッドにより,下記の95成分+2-Isopropylmalic acidの一斉分析が可能です。
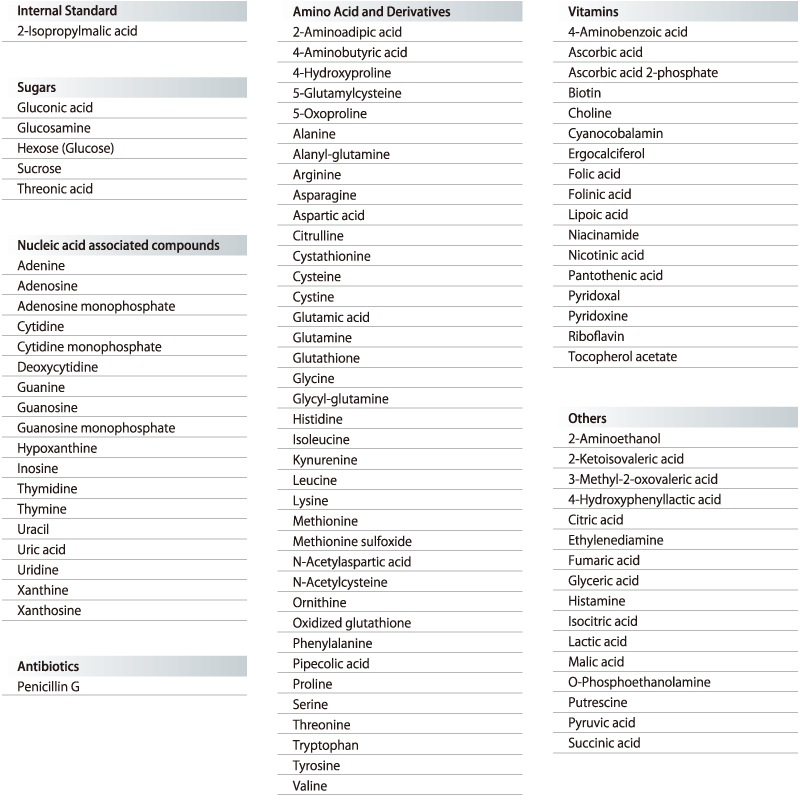
下記の培地,培地添加物についてC2MAPシステムが適応可能であることを確認しています。
| 細胞種 | CHO細胞 | iPS/ES細胞 | T細胞 | 間葉系幹細胞 |
|---|---|---|---|---|
| 培地 | BalanCD® CHO | AK03N | X-VIVO™ 10 | MSCBM |
| 1×CD CHO | Essential-8™ | X-VIVO™ 15 | MesenPRO™ | |
| EX-CELL® CHO | mTeSR™1/TeSR™-E8™ | TexMACS™ | Stempro® | |
| 添加物 | Fetal Bovine Serum(100% v/v) | |||
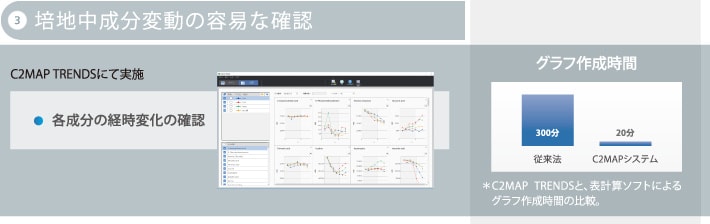
培地中成分変動の容易な確認
- 得られた各成分の経時変化をトレンドグラフとして表示することが可能
- 複数の実験条件の結果を重ねて表示することで、比較解析が可能
 C2MAP TRENDS
C2MAP TRENDS
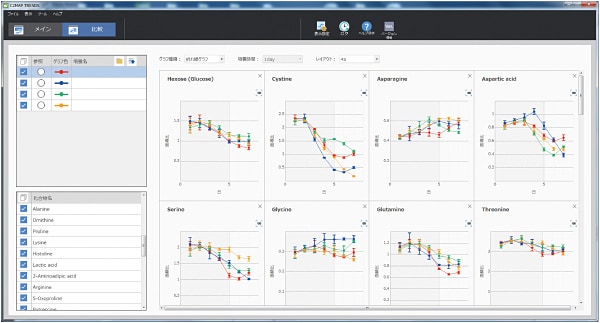
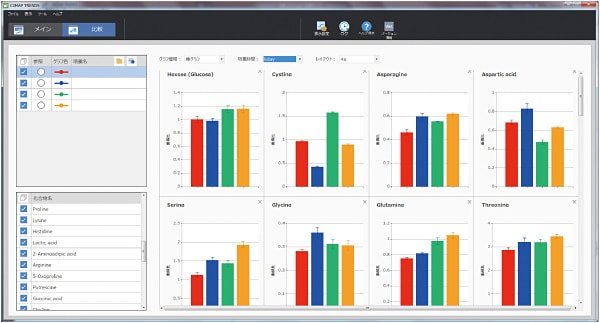
LC/MS/MSによって得られた結果は,専用のViewerソフトにより,成分毎の時間変動をグラフ化することができます。培養日数ごとの培地成分および細胞分泌代謝物の変動モニターや,異なる培養系列試料での成分比較などのグラフを表示することで,培地成分の消費や枯渇,細胞分泌代謝成分量の増減を見ることがき,最適な培養条件の検討や,細胞状態を把握するための有用な知見を提供します。
◆測定対象成分毎の経時変化
◆異なる培養系列試料間での測定対象成分比較
柔軟なシステム構成に対応
- 自動前処理部を切り離して設置することで、前処理とLC/MS/MS測定を別室で行うことが可能
オペレーターに依存せず,高品質の測定データが得られる細胞培地分析
LC/MS/MSトータルシステム

自動前処理に必要なモジュールのみをLC/MS/MSと切り離して稼働可能

- LC/MS/MSは前処理部分とは別室に設置して,LC/MS/MSを他の分析と共用することが可能です。
- 培養室から培養サンプルを持ち出せない場合,サンプル前処理を培養室で実施し,別室のLC/MS/MSで測定することが可能となります。
- 自動前処理部分を切り離すことにより,実験室の省スペース化が可能です。
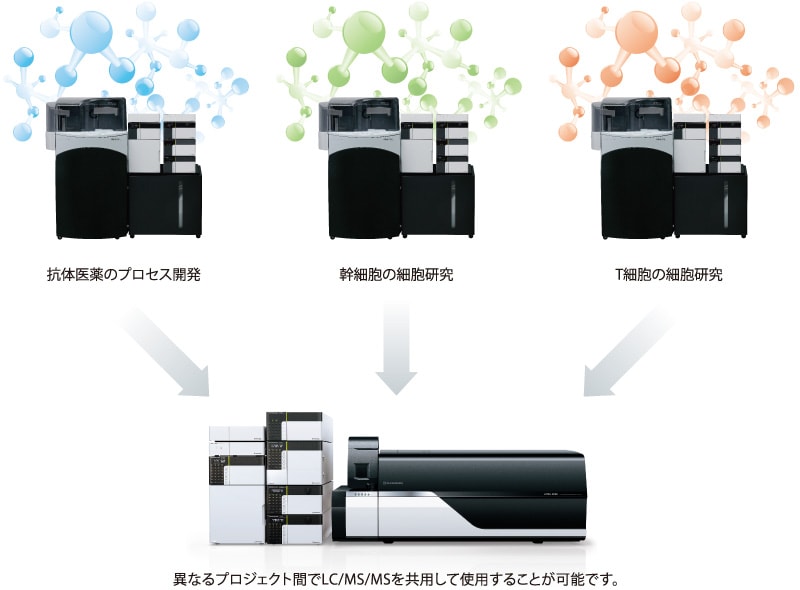
システム構成の柔軟性が生み出す新たな価値
サンプルの自動前処理部分とLC/MS/MS部分を異なる実験室に設置して使用することができます。例えば,培養室においてサンプルの前処理までを行い,分析室に設置された共用のLC/MS/MSにて分析を行うということも可能です。

※培養室からのサンプル持ち出しは,各社の規定に沿って行ってください。

最適なワークフローが選択可能
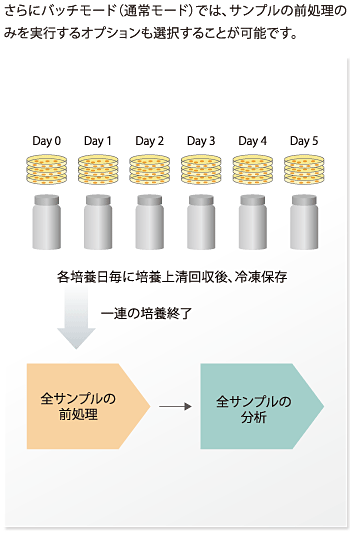
C2MAPシステムでは,セットした培養上清サンプルの前処理を優先してから行うバッチモード(通常使用を想定)と,各サンプルごとに前処理と分析を交互に行う逐次モードの両方に対応しています。
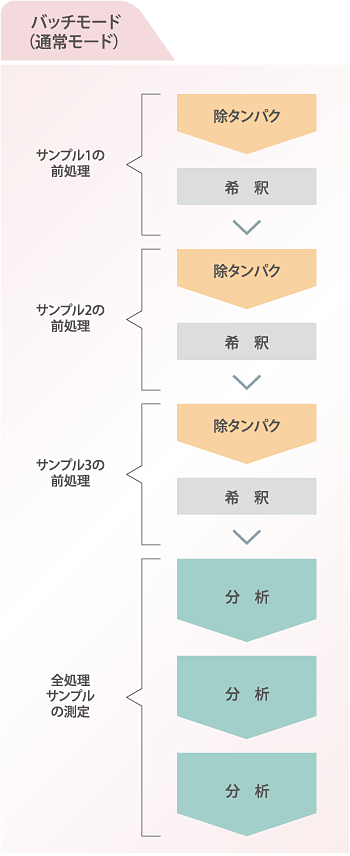
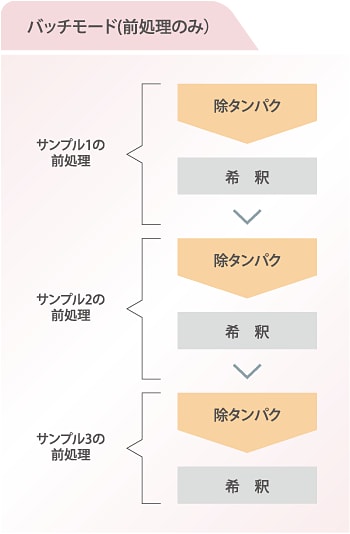
バッチモード(通常モード)では,サンプルの除タンパクを優先して実行するため,サンプル中に存在する酵素タンパク質の不活化が可能で,サンプルの安定性向上を図ることができます。
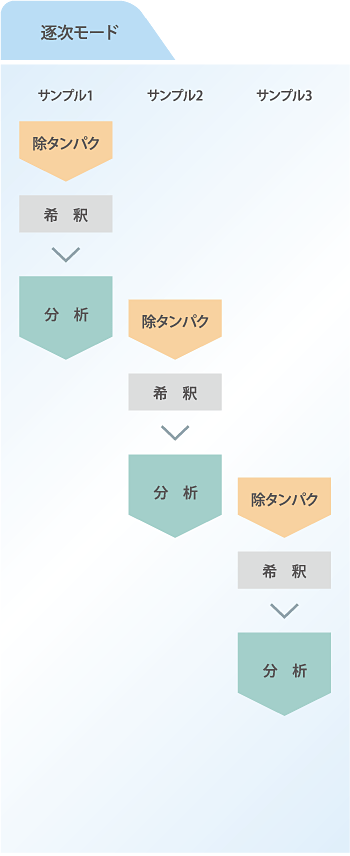
逐次モードではLC/MS/MS測定とC2MAP-2030での除タンパク操作が並行して実施されるので,総分析時間の短縮が可能です。