定性関連操作,ライブラリ関連操作
| Q: | 手動波形処理を実行したい①(お問い合わせ番号0623) | |||||||||
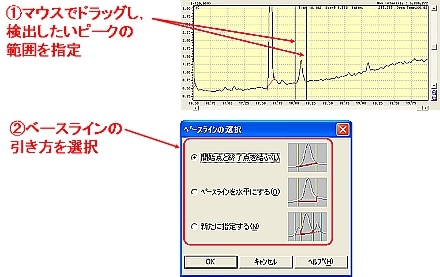
| A: | 微小な信号強度のピークに対し波形処理パラメータを初期値として自動波形処理を行い,処理がうまく行われなかった場合,手動波形処理を利用すると効果的です。 ここでは,TICにおける未検出ピークを手動波形処理にて検出させる方法を紹介します。
|
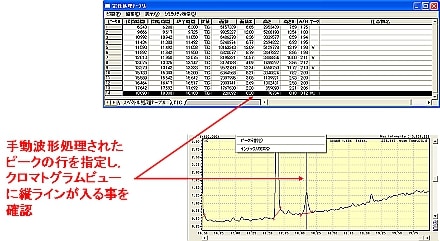
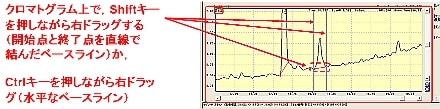
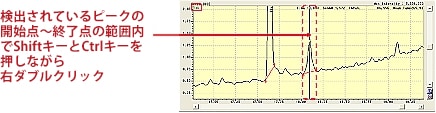
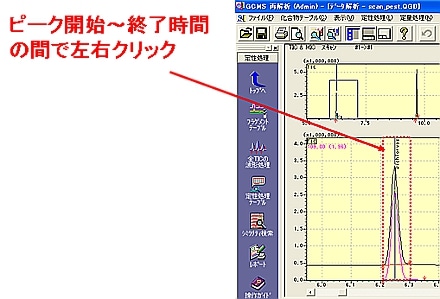
| Q: | 手動波形処理を実行したい②(キーボードのキーとマウス操作の組み合わせ) (お問い合わせ番号0624) | ||||||||
| A: |
ここでは,キーボードとマウスを活用した手動波形処理の方法についてご紹介します。
|
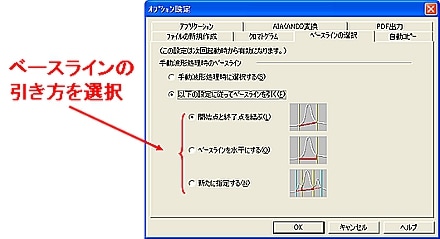
| Q: | 手動波形処理のベースラインの引き方を固定したい(お問い合わせ番号0625) | |||
| A: | 手動波形処理のベースライン設定は1つに決めており変更の必要がない場合,設定を固定すると,手動波形処理を行うごとにベースラインを選択する手間が省けます。
|
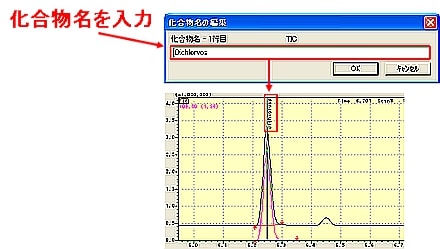
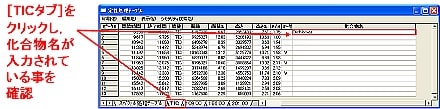
| Q: | 定性ピークテーブルの化合物名の入力をクロマトグラムビュー上で行う(お問い合わせ番号0626) | |||||||
| A: |
多数の化合物ピークがあるクロマトグラムのピークトップコメントとして,化合物名が表示されているとピークの判別が容易です。
|
| Q: | 表示スペクトルを標準スペクトルとして登録したい(お問い合わせ番号0601) | ||||||
| A: |
|
| Q: | ライブラリのスペクトルを標準スペクトルとして登録したい(お問い合わせ番号0602) | ||||||||
| A: |
|
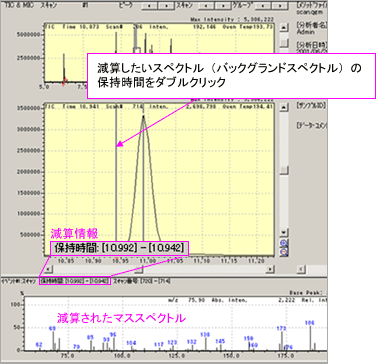
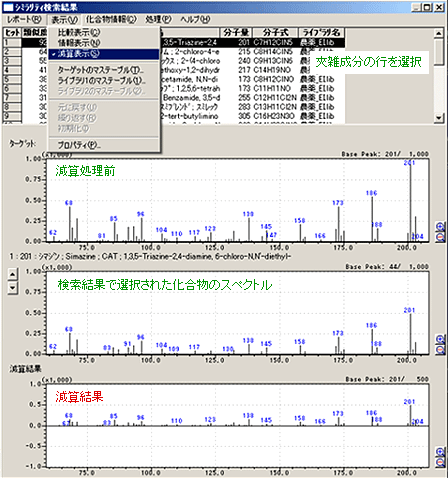
| Q: | スペクトルの減算処理したい(ノイズの除去)(お問い合わせ番号0615) | |||||
| A: |
マススペクトル測定で得られるデータには,カラムの夾雑成分によるバックグラウンドや残留ガスのピークが含まれています。 またキャピラリーカラムでも,ピークを完全分離できない時に隣接した成分のピークが目的成分とオーバラップし定性を難しくすることがあります。このようなときにマススペクトルのバックグランド減算処理を行うと,より正確な定性ができます。
|
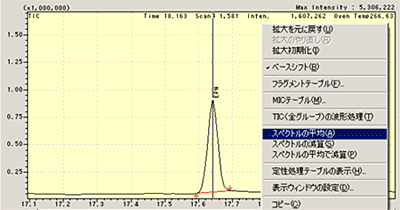
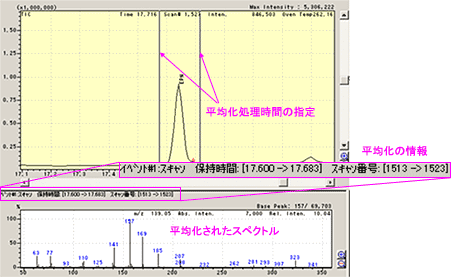
| Q: | マススペクトルの平均化処理をしたい(お問い合わせ番号0620) | ||||||
| A: | スキャンモードでマススペクトル測定をする場合,1サイクルのスペクトルを採取するのに設定されたスキャンインターバルに応じてある程度の時間がかかります。 一方クロマトグラムは刻一刻と変化しているので,低質量側の強度と高質量側の強度との比は,測定されたスペクトルと実際のスペクトルとでは若干異なってきます。 このような場合,ピークトップを含めたマススペクトルの前後を平均化処理することにより,より正確なマススペクトルにすることができます。
|
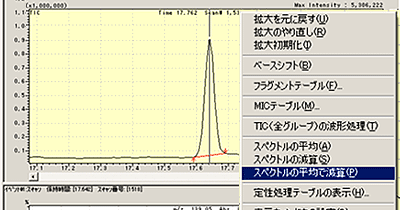
| Q: | スペクトルの平均で減算処理をしたい(お問い合わせ番号0621) | |||||||
| A: | スペクトルの平均での減算処理にてノイズ成分のマススペクトルを除去し,より正確なシミラリティ検索を行う方法を紹介します。
|
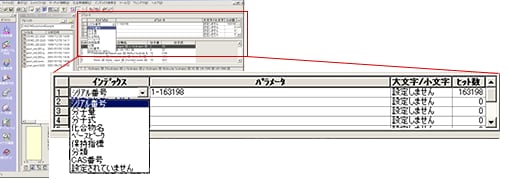
| Q: | インデックス検索をしたい(お問い合わせ番号0611) | ||||||||||
| A: |
ライブラリファイル内から,特定の検索インデックスを対象にマススペクトルを選び出すインデックス検索は,所有のライブラリファイルにその化合物が登録されているかどうかの確認や,ライブラリに含まれる化合物の詳細な情報を知りたい場合に有効です。
|
||||||||||
|
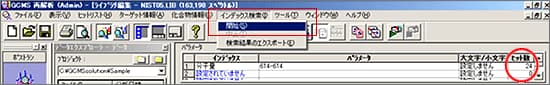
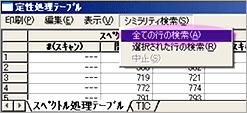
| Q: | 複数のピークをまとめてシミラリティ検索したい(お問い合わせ番号0616) | |||||
| A: |
検索対象のピークが複数の場合,ピークを一つずつ選択しシミラリティ検索を行うのは手間がかかりますが,TIC波形処理で複数の目的成分のピーク検出を行い,検出された複数のピークをシミラリティ検索できます。
|
|||||
|
| Q: | 条件を絞ってシミラリティ検索したい(ポストサーチ)(お問い合わせ番号0617) | |||
| A: |
検索インデックスを設定して,ライブラリの検索対象データをある範囲に限定し,シミラリティ検索の結果を絞るインデックス検索をポストサーチといいます。 予め未知化合物の分子量が大体分かっている場合や,炭素数が分かっている場合などにポストサーチを利用することができ,下記条件で絞込みできます。
|
|||
|
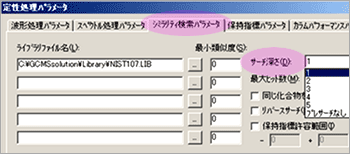
| Q: | より詳しいシミラリティ検索したい(サーチ深さ)(お問い合わせ番号0618) | |||
| A: |
プレサーチは,未知試料のスペクトルとライブラリ登録スペクトルの簡単な比較を行い,明らかに2つのデータに相違がみられる場合にはそのライブラリ登録スペクトルを検索対象から除外するというフィルターの役目をします。
|
|||
|
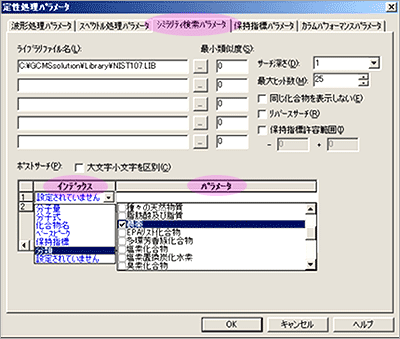
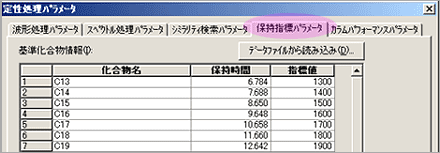
| Q: | 保持指標を用いたシミラリティ検索したい(お問い合わせ番号0619) | ||||||
| A: |
シミラリティ検索を行う時,類似度で検索した結果から,さらに保持指標の範囲で絞込みを行うかどうかを指定することができます。保持指標を利用すると,よりいっそう確実な成分同定が可能です。例えば,異性体などスペクトルが類似した成分の分析(香料分野)や高マトリックス中での多成分分析(食品分野)の定性機能の強化ができます。
|
||||||
|
| Q: | シミラリティ検索結果を印刷したい(お問い合わせ番号0622) | ||
| A: |
|
||
|
| Q: | 夾雑物を避けた検索をしたい(リバースサーチしたい)(お問い合わせ番号0603) | ||||||
| A: |
リバースサーチでは,ライブラリに登録されているイオンピークの質量(50個)にのみ注目して類似度の計算を行います。 元のスペクトルのピークから,ライブラリスペクトルに現れている質量のものだけを抽出し,それに対して類似度の計算を行います。 ライブラリスペクトルに含まれていない質量のピークは無視されます。 マススペクトルに夾雑物が混じっていたり,ノイズが多い場合に有効です。
|
| Q: | 減算スペクトルを再検索をしたい(マルチスペクトル処理)(お問い合わせ番号0612) | |||||||
| A: |
ターゲットスペクトルが複数の成分と重なっているとわかっている場合,シミラリティ検索してヒットしたスペクトルの中から任意のスペクトルを減算し,再度シミラリティ検索することで,より確実なシミラリティ検索結果を得ることができます。 その減算結果を再度シミラリティ検索することもできます。 (夾雑成分のスペクトルを複数回減算する機能を,マルチスペクトル処理といいます。)
|
| Q: | デフォルトライブラリを設定したい(お問い合わせ番号0613) | |
| A: | シミラリティ検索パラメータ中で検索対象となるマススペクトルライブラリ名が登録されていないときに,デフォルトライブラリファイルと最小類似度がシミラリティ検索で使用されます。 [GCMS再解析]ウィンドウ データ解析画面で,[ツール]メニューの[デフォルトライブラリの設定]を選択して,[デフォルトライブラリの設定]画面を開きます。 [...]ボタンをクリックして,ライブラリサーチに用いるMSスペクトルライブラリファイルを選択します。 最高5ファイルまで選択できますが,1ファイルでもかまいません。 ライブラリ検索時,未知スペクトルは,設定した全てのライブラリファイル内から検索されます。 検索結果として出力させるライブラリスペクトルの最低類似度を,ライブラリファイルごとに設定します(設定範囲:0-99)。 設定後,[OK]ボタンで閉じます。
|
| Q: | 試料中で目的の化合物に最も類似度の高いピークを検出したい(コンパウンドファインダーしたい)(お問い合わせ番号0604) | ||||||||||
| A: |
コンパウンドファインダーとは,試料中で目的の化合物に一番類似度の高いピークを検出する機能です。 質量情報を化合物テーブルに登録しておけば,採取データから標準スペクトルと類似度(SI)の高いスペクトルもつピークを探し出します。
|
| Q: | 測定データからオリジナルのライブラリを作りたい(化合物を1つずつ登録)(お問い合わせ番号0605) | ||||||||||
| A: |
自分だけのライブラリを作成したい場合やパブリックライブラリ(NISTやWileyなど)に登録されていない化合物をオリジナルライブラリに新規に登録したい場合に,下記方法が利用できます。 データファイルに保持指標パラメータが登録されていれば,保持指標が入力されたライブラリを作成することができます(ただし,n-アルカンなどの基準化合物のデータファイルが必要です。)。 |
||||||||||
|
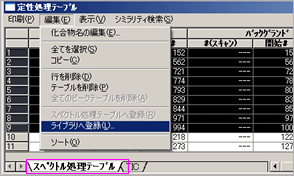
| Q: | 測定データからオリジナルのライブラリを作りたい(複数の化合物を登録する)(お問い合わせ番号0614) | |||||||||||
| A: | スペクトル処理テーブルから複数の化合物をライブラリに登録できます。 複数の化合物を登録すると,処理が簡便なだけではなく保持指標が自動的に登録されます。
|
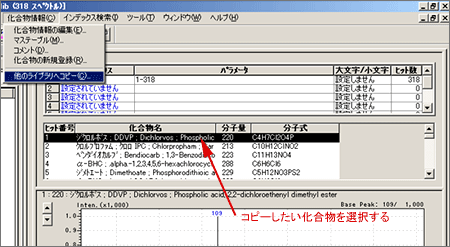
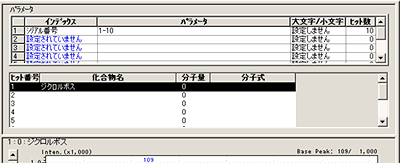
| Q: | パブリックライブラリに登録されている化合物をオリジナルライブラリに登録したい(コピーしたい)(お問い合わせ番号0606) | ||||||||
| A: |
|
||||||||
|
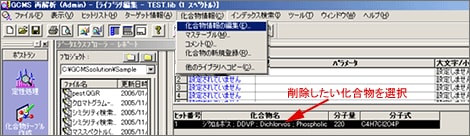
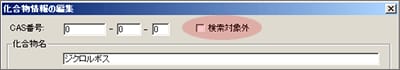
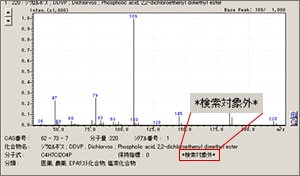
| Q: | オリジナルライブラリに登録した化合物を削除したい(お問い合わせ番号0607) | |||||||||
| A: |
|
| Q: | 構造式の無いライブラリへ構造式を登録したい(お問い合わせ番号0608) | |||
| A: |
NISTやWileyなどパブリックライブラリには構造式ファイルが添付されているので,画面やレポートに構造式が出力されますが,オリジナルライブラリは構造式ファイルを添付できません。
|
|||
|
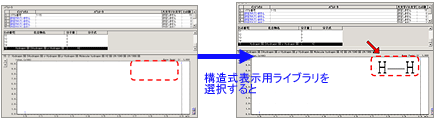
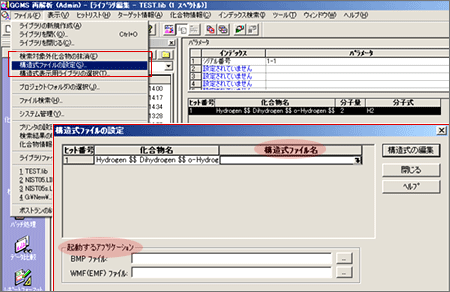
| Q: | 画像ファイルからオリジナルライブラリに構造式を登録したい(お問い合わせ番号0609) | |||
| A: |
オリジナルライブラリを使う場合は,各化合物に対応する構造式ファイルをライブラリファイルに登録できます。 ビットマップファイル(*.BMP)あるいはウィンドウメタファイル(*.WMF)から選択できます。 登録するには事前に構造式の画像ファイルが必要です。
|
|||
|
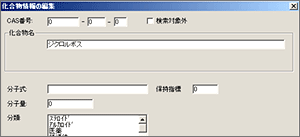
| Q: | ライブラリに保持指標を編集/入力したい(お問い合わせ番号0610) | ||
| A: |
GCMSsolutionでは,保持指標が登録されているライブラリを使用することにより,保持指標でさらに絞り込み検索を行い,高精度な成分特定をすることも可能です。
|
||
|



![[定性処理テーブル]の[TIC]タブ](/sites/an.shimadzu.co.jp/files/ckeditor/gcms/support/faq/gcmssol/fig170.gif)
![[定性処理パラメータ]画面の[保持指標パラメータ]タブ](/sites/an.shimadzu.co.jp/files/ckeditor/gcms/support/faq/gcmssol/fig174.gif)
![[定性処理パラメータ]画面の[シミラリティ検索パラメータ]タブ](/sites/an.shimadzu.co.jp/files/ckeditor/gcms/support/faq/gcmssol/fig175.gif)
![[データ解析]画面](/sites/an.shimadzu.co.jp/files/ckeditor/gcms/support/faq/gcmssol/fig204.gif)
![[シミラリティ検索結果]画面](/sites/an.shimadzu.co.jp/files/ckeditor/gcms/support/faq/gcmssol/fig205.gif)

![[定性処理パラメータ]画面](/sites/an.shimadzu.co.jp/files/ckeditor/gcms/support/faq/gcmssol/fig60.gif)
![[定性処理パラメータ]画面](/sites/an.shimadzu.co.jp/files/ckeditor/gcms/support/faq/gcmssol/fig61.gif)
![[定性処理テーブル]画面](/sites/an.shimadzu.co.jp/files/ckeditor/gcms/support/faq/gcmssol/fig154.gif)
![[化合物情報の編集]画面](/sites/an.shimadzu.co.jp/files/ckeditor/gcms/support/faq/gcmssol/fig69.gif)